Biometrische Methodenforschung
Unsere Aktivitäten im Bereich der biometrischen Methodenforschung sind von anwendungsbezogenen Fragestellungen motiviert und umfassen folgende Schwerpunkte
Fallzahlkalkulation und -rekalkulation
(M. Kieser, M. Kirchner, A. Sander)
Adaptive Designs und multiple Testprozeduren
(M. Kieser, A. Sander)
(M. Kirchner, M. Kieser, T. Byczkowski)
(L. Baumann, M. Kieser, L. Sauer, P. Thalmann)
(M. Kieser)
Multiple IMputation In CLInical Prediction modelling (MIMICLIP)
(M. Feisst, M. Kieser, S. Awounvo)
Automating Screening in Systematic Reviews
(J. Vey, M. Kieser)
Robust statistical methods for genetic-molecular studies
(J. Lorenzo Bermejo, D. Scherer)
Fallzahlkalkulation und -rekalkulation
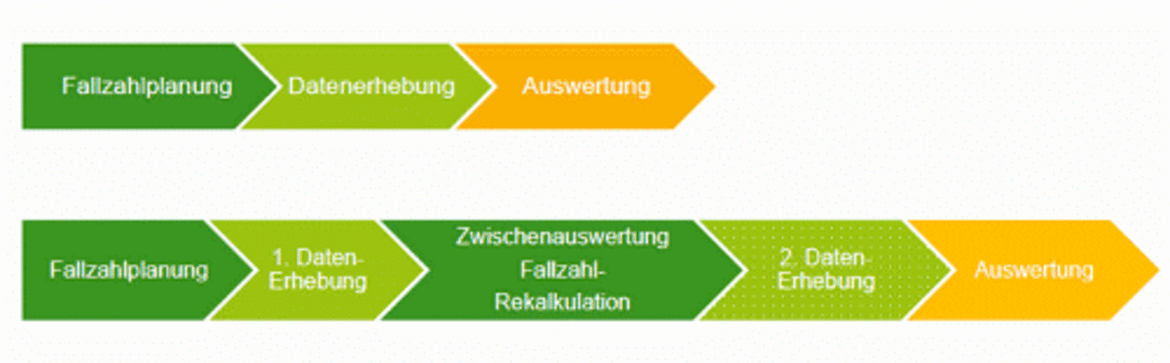
Die Berechnung der erforderlichen Fallzahl ist in klinischen Studien immer dann eine besondere Herausforderung, wenn für die Schätzung der Parameter keine ausreichende Basis in der Literatur existiert. Eine valide Schätzung dieser Parameter vor Beginn einer Studie ist dann sehr schwierig und birgt die Gefahr, die benötige Fallzahl der Studie deutlich zu über- oder zu unterschätzen. Eine Möglichkeit, diese Fehlspezifikation zu korrigieren, bietet ein Studiendesign mit interner Pilotstudie. Im Rahmen der internen Pilotstudie werden Störparameter verblindet neu geschätzt und die benötigte Fallzahl entsprechend angepasst.
Adaptive Designs ermöglichen die Modifikation des Stichprobenumfangs basierend auf den Ergebnissen entblindeter Zwischenauswertungen.
Arbeitsgruppe Klinische Studien
Adaptive Designs und multiple Testverfahren
Adaptive Designs erlauben, unter Kontrolle des Signifikanzniveaus, in einer laufenden Studie Veränderungen am Studiendesign vorzunehmen. Die Möglichkeiten von Design-Änderungen gehen dabei weit über eine Modifikation des ursprünglich festgelegten Stichprobenumfanges hinaus. Beispielsweise können die zu untersuchenden Behandlungsgruppen, die Patientenpopulation, die Zielgröße oder der für die Auswertung vorgesehene statistische Test auf Basis der verfügbaren Daten abgeändert werden, wenn sich die Planungsannahme, auf der die entsprechende Festlegung ursprünglich basierte, im Studienverlauf als fehlerhaft herausstellt.
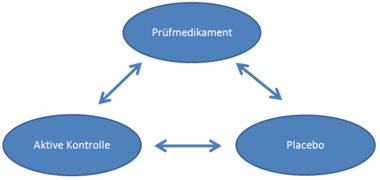
Multiple Testverfahren werden angewendet, um die Wahrscheinlichkeit eines Fehlers erster Art zu kontrollieren, wenn mehrere Vergleiche in derselben Stichprobe durchgeführt werden. Aktuelle Guidelines empfehlen beispielsweise, Nichtunterlegenheits-Studien nach Möglichkeit in einem dreiarmigem Studiendesign mit dem Prüfmedikament, einer aktiven Kontrolle und einer Placebo-Gruppe durchzuführen. Der Nichtunterlegenheits-Nachweis des Prüfmedikaments gegenüber der aktiven Kontrolle und der Nachweis der Überlegenheit des Prüfmedikaments und der aktiven Kontrolle gegenüber Placebo führen in diesem Studiendesign zu einem multiplen Testproblem.
Arbeitsgruppe Klinische Studien
Consideration of missing values in sample size calculation and re-calculation for clinical trials (COMIVA)
Determination of the appropriate sample size to achieve high probability to detect a clinically relevant treatment effect is a crucial aspect when planning a clinical trial. Both, the choice of a too small or a too high sample size is to be criticized for ethical, scientific, and economic reasons. A problem that frequently occurs is that some data are missing so that outcome data important for the statistical analysis were not collected. Missing data may lead to a power decrease as the sample size of the dataset used for statistical analysis is smaller. Therefore, the possibility of missing data has to be considered already during the planning phase of the clinical trial. A common way to deal with the issue of missing data during sample size calculation is the inflation of the sample size to account for the expected drop-out rate. However, all included patients are to be considered in the analysis based on the intention-to-treat principle. Thus, imputation methods are typically used to replace missing outcome data. As a consequence, treating patients with missing data in the sample size calculation as non-existent for the analysis does not reflect the actual situation. This project contributes to the improvement of sample size determination for a clinical trial that uses imputation methods in the primary analysis to replace missing outcome data with the expectation that the required sample size can be reduced.
This project is funded by the German Research Organization (DFG), grant KI 2109/1-1
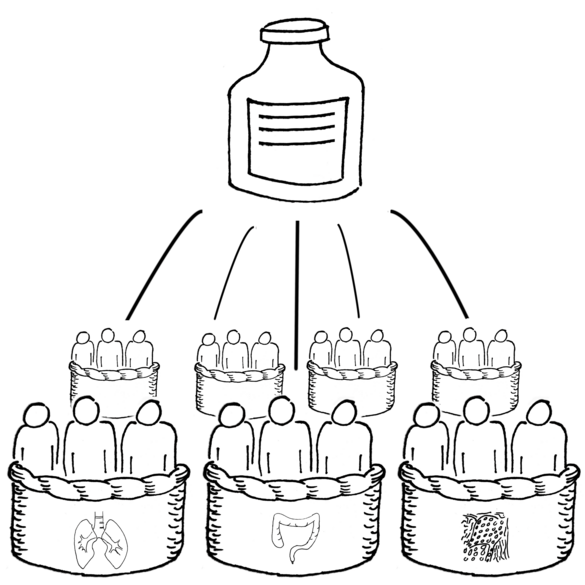
Basket trials
Basket trials investigate the efficacy of a new treatment simultaenously in several patient subgroups. They are mostly applied in oncology, where the subgroups comprise patients with different tumour histologies but all patients share a common genetic mutation. The rationale of these trials is therefore that the genetic mutation is more important for the efficacy of the drug than the histology.
Naïve approaches for the analysis of basket trials are to either analyse each subgroup individually or to pool the data of all baskets before the analysis is conducted. However, individual analyses often lack sufficient power while a pooled analysis ignores that the treatment may only be effective in a subset of the included histologies. Bayesian methods such as hierarchical modelling and model averaging offer a middle ground, as they allow partial pooling of data based on the observed heterogeneity between subgroup effects. While these methods are superior to the naïve approaches in terms of important operating characteristic such as type 1 error rate and power, they are computationally very expensive.
At the IMBI, we develop new and extend existing basket trial designs with a focus on designs that utilise computationally cheaper empirical Bayes methods. Another focus is the development of user-friendly R packages.
Onkologische Phase-II-Studien
In klinischen Studien der Phase II wird zum ersten Mal die Wirksamkeit einer erfolgversprechenden Therapie an Patienten untersucht. Auf Basis der Studienergebnisse wird entschieden, ob das Entwicklungsprogramm abgebrochen wird oder das Nutzen-Risiko-Profil aussichtsreich genug ist, um die Durchführung von wirksamkeitsbeweisenden Phase-III-Studien mit einer großen Patientenzahl zu rechtfertigen. In der Onkologie werden Phase-II-Studien aus ethischen und ökonomischen Gründen in aller Regel einarmig und mit geplanter Zwischenauswertung durchgeführt, um eine möglichst frühzeitige Entscheidung für oder gegen eine Fortsetzung der Studie zu ermöglichen. Im Rahmen eines durch die Deutsche Forschungsgemeinschaft (DFG) geförderten Projektes wurden für diese Anwendungssituation Studiendesigns entwickelt, die datengesteuerte Modifikationen von Design-Charakteristika (z.B. der Fallzahl) ermöglichen, wobei die Kontrolle des Fehlers 1. Art zu einem vorgegebenen Signifikanzniveau sichergestellt ist. Das Projekt wird seit April 2014 für weitere drei Jahre von der DFG weitergefördert.
Weiterhin wurde am IMBI ein benutzerfreundliches statistisches Software-Tool entwickelt, welches die Planung, das statistische Monitoring und die Auswertung onkologischer Phase-II-Studien unterstützt. Die Umsetzung des Programms wurde durch das Bundesministerium für Bildung und Forschung (BMBF; Förderkennzeichen 01EZ1206) finanziell gefördert.
Eine Kurzbeschreibung des Software-Tools finden Sie hier
Die zugehörige Publikation mit Sourcecode finden Sie hier.
Eine Internet-App zur Berechnung optimaler und flexibler zweistufiger Phase-II-Designs (auch bei over- und under-running der ursprünglich geplanten Fallzahl in einer der beiden Stufen) finden Sie hier
Multiple IMputation In CLInical Prediction modelling (MIMICLIP)
Multiple imputation is a powerful and flexible technique for handling missing data in predictive modeling. Its ability to account for uncertainty and preserve the variability in the data makes it a valuable tool in research and practice. However, the implementation of multiple imputation requires careful consideration and validation, especially regarding the timing of the imputation in the model development and validation process. It is currently unknown at which specific time point multiple imputation should be employed in the development of a clinical prediction model. This project will help establish clearer guidelines on this matter, ultimately leading to more standardized practices in clinical prediction model development.
This project is funded by the German Research Organization (DFG), grant FE 2364/2-1
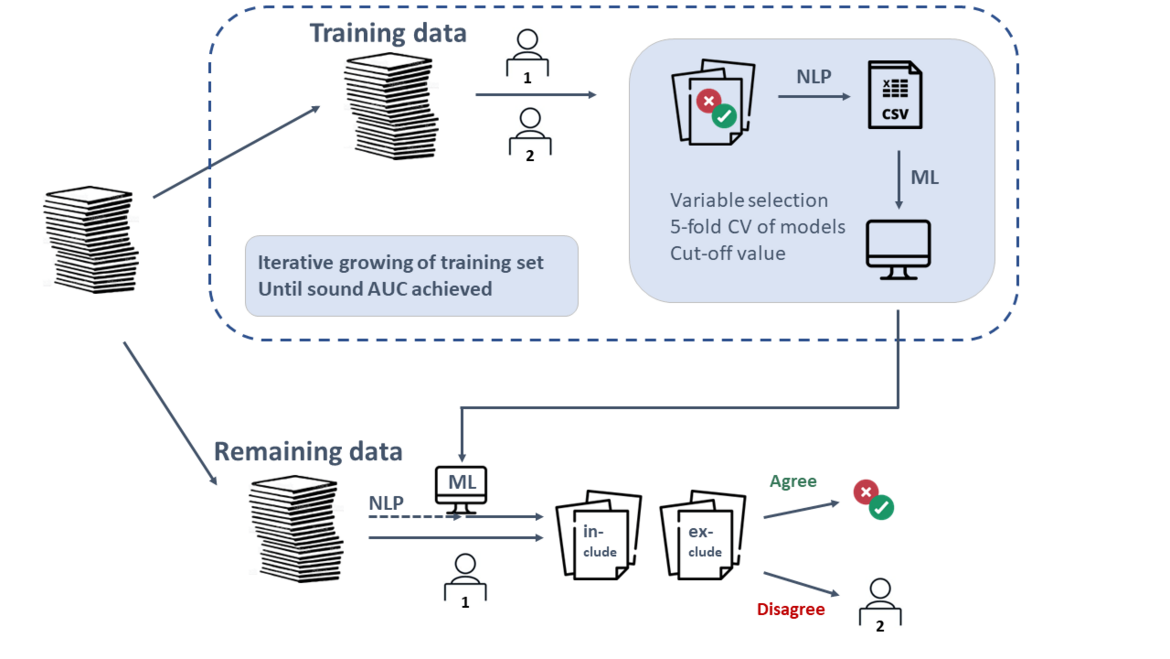
Automating Screening in Systematic Reviews
The screening process in systematic reviews is extremely time- and cost-intensive, as a large number of studies often need to be manually reviewed and assessed for relevance. The use of machine learning methods and large language models (LLMs) offers the potential to partially automate this process. As a result, a reviewer can be replaced in certain steps, allowing for savings in both time and human resources.
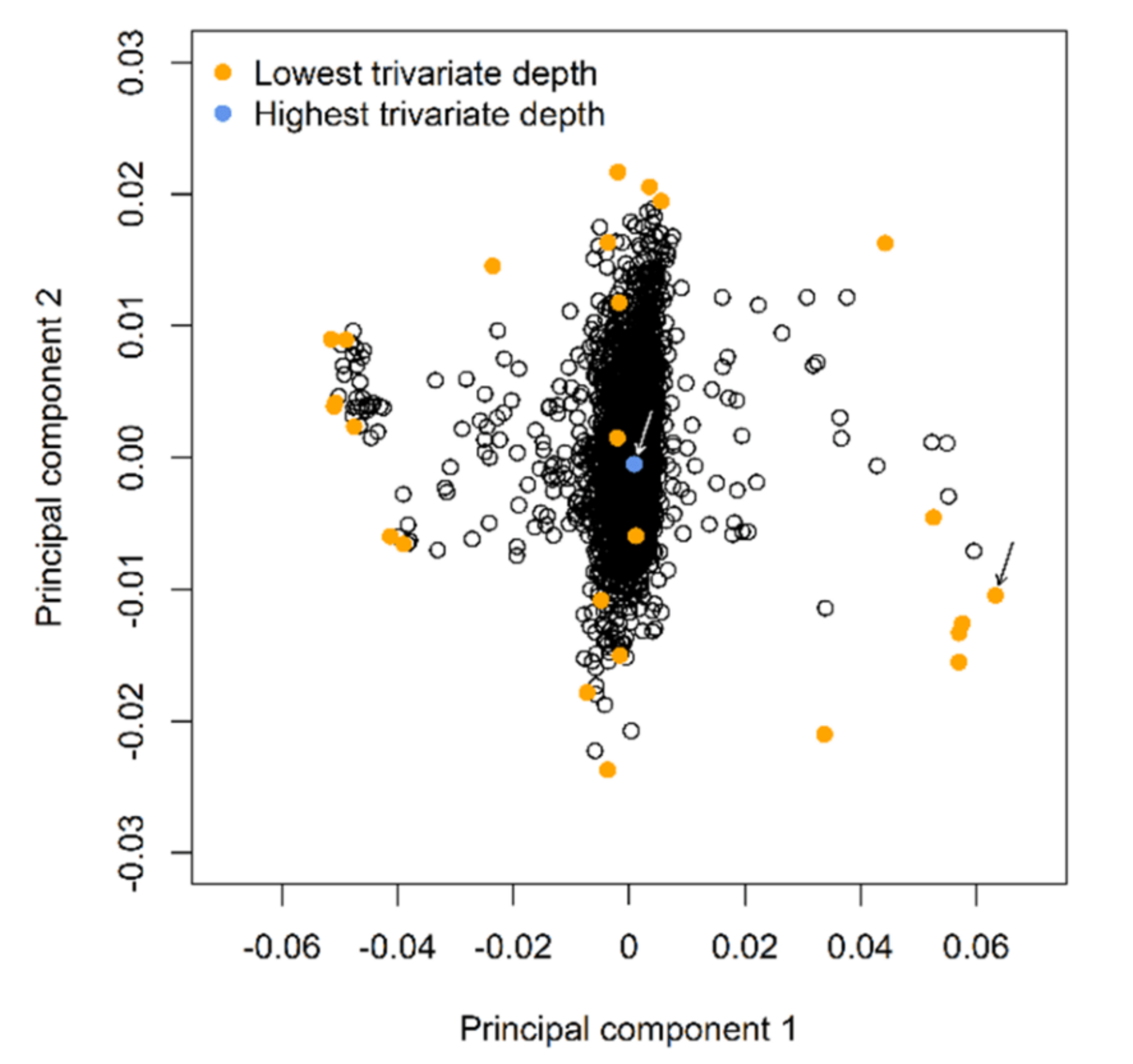
Robust statistical methods for genetic-molecular studies
LASSO (Least absolute shrinkage and selection operator) regression is often used to select the most promising single nucleotide polymorphisms (SNPs) associated with a specific molecular phenotype. While the penalty parameter λ restricts the number of selected SNPs and the potential model overfitting, the least-squares loss function of standard LASSO regression leads to a strong dependence of statistical results on a small number of individuals with phenotypes or genotypes that deviate from the majority of the study population—typically comprised of outliers and high-leverage observations. We are conducting simulations and analyzing real genotype, methylation, protein, metabolite, small-RNA and mRNA expression data to compare the stability of penalization, cross-iteration concordance, false-positive and true-positive rates, and the prediction accuracy of robust LASSO regression methods such as the robust Huber-LASSO.