- Biobank / GMMG Probeneingangslabor
- Myelomregister
- Myelom Studienzentrale / GMMG Studien
- Translationale Myelomforschung
- Funktionelle Bildgebung
- Molekularbiologisches Labor / Studien zur Erblichkeit des Multiplen Myeloms
- Innovative Therapien beim Multiplen Myelom: Phase I, PerMyt, HIPO
- Laboratory for Translational Immunology (TRIM)
- GMMG Assessment Center der Minimalen Resterkrankung (MRD)
Molekularbiologisches Labor / Studien zur Erblichkeit des Multiplen Myeloms
Die vererblichen, genetischen Risikofaktoren vieler Krebserkrankungen sind ein aktuelles Thema der Krebsforschung. Trotz der Identifikation zahlreicher genetischer Veränderungen, die mit dem Erkrankungsrisiko für verschiedene Krebsarten assoziiert werden konnten, sind die Kenntnisse hinsichtlich vererblicher Risikofaktoren noch lückenhaft. Für die Erklärung von familiären Häufungen des Multiplen Myeloms sind die bis heute identifizierten genetischen Veränderungen jedoch nicht ausreichend. Aktuelle Forschungsergebnisse deuten darauf hin, dass es noch zahlreiche unbekannte genetische Einflussfaktoren gibt, die Aussagen über den Therapieerfolg oder die Prognose zum Verlauf der Myelom-Erkrankung erlauben könnten. Vergleichsanalysen zwischen der genomischen DNA von Patienten und der DNA von gesunden Vergleichspersonen könnten solche bisher unbekannte genetische Varianten aufdecken und so die Früherkennung und Therapie des Multiplen Myeloms verbessern.
Gleichzeitig zum Multiplen Myelom erforschen wir auf gleiche Weise auch eng verwandte Vorläufererkrankungen wie das Smouldering Multiple Myelom (SMM) und Monoklonale Gammopathie unklarer Signifikanz (MGUS). Außerdem untersuchen wir in Kooperation mit dem Amyloidose Zentrum am Universitätsklinikum Heidelberg die mit dem Multiplen Myelom oder MGUS assoziierte Amyloidose.
Family Sequencing Projekt

Im Rahmen eines durch die International Myeloma Foundation (IMF) mit einem „Black Swan Research Initiative“ Grant geförderten Projekts untersuchen wir mittels Next Generation Sequencing Familien, in denen es gehäuft zu Erkrankungen am Multiplen Myelom gekommen ist.
Im Zuge des Projektes wird die DNA aller Teilnehmer - jeweils nach Familienzugehörigkeit eingeordnet. Es werden genetische Varianten untersucht, die ausschließlich in den erkrankten Familienmitgliedern zu finden sind, nicht aber in den gesunden Verwandten. Eine solche Vergleichsanalyse könnte bisher unbekannte genetische Varianten aufdecken, die für die Entstehung des Multiplen Myeloms verantwortlich sind.
Genomweite Assoziation Studien
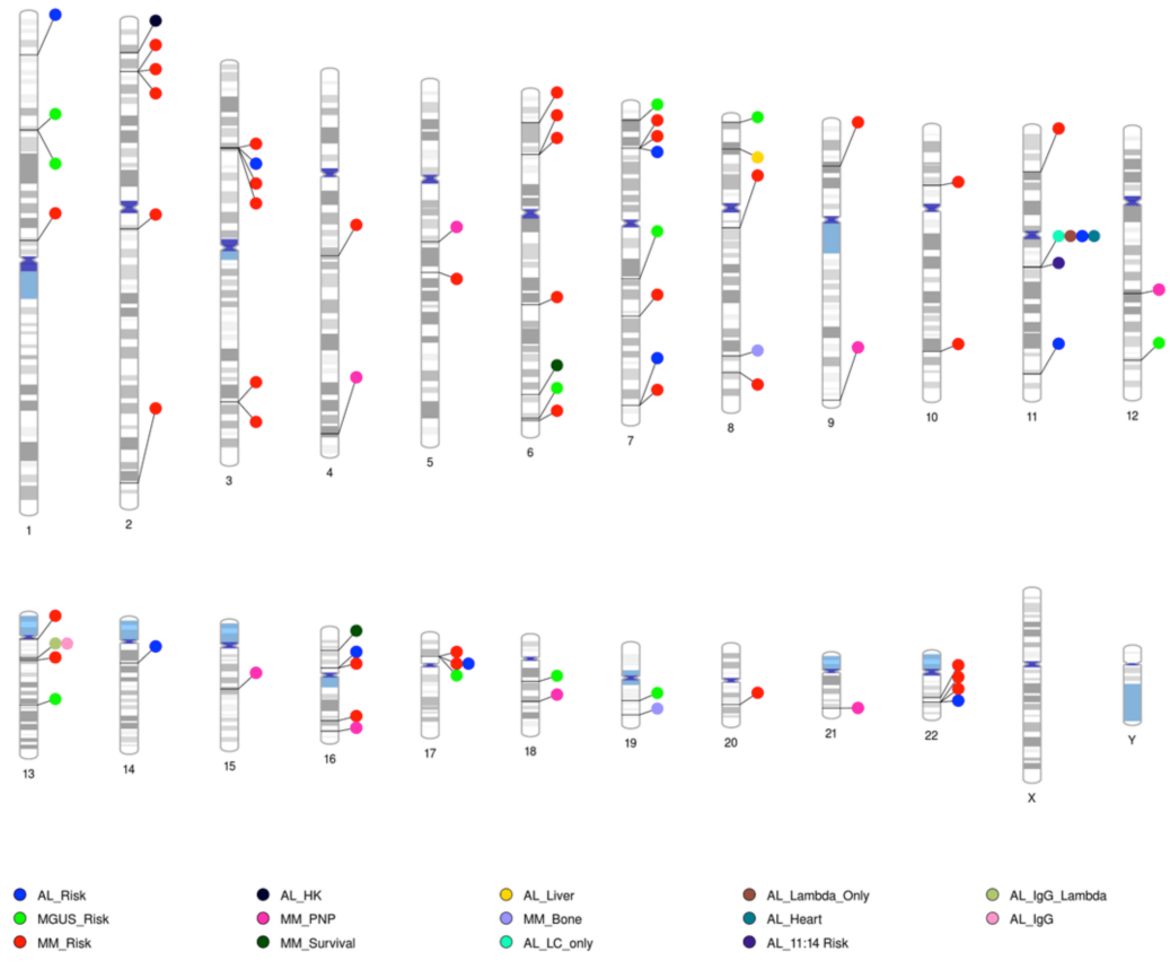
Chromosomale Regionen an denen genetische Varianten für das Risiko von MM, AL und MGUS identifiziert werden konnten.
Neben dem Family Sequencing Projekt führen wir in enger Zusammenarbeit mit dem Deutschen Krebsforschungszentrum epidemiologische, genetische Assoziationsstudien zur Erforschung genetischer Risiko-Varianten durch, die in der Bevölkerung mit einer relativen Häufigkeit von >10% auftreten können. Hierfür vergleichen wir mittels „genomweiter Genotypisierung“ (GWAS) das Erbgut einer Vielzahl nicht verwandter erkrankter und gesunder Studienteilnehmer, um diejenigen Erbgutveränderungen zu finden, die ein erhöhtes genetisches Grund-Risiko verantworten am Multiplen Myelom zu erkranken. Neben dem genetisches Grund-Risiko erforschen wir auch, ob vererbliche genetische Varianten mit einer bestimmten klinischen Form des Myeloms oder einer bestimmten Prognose assoziiert sind. So konnten wir z.B. zeigen, dass genetische Varianten auf den Chromosomen 4 und 5 mit einem erhöhten Risiko verbunden sind, unter Bortezomib Therapie eine Polyneuropathie zu entwickeln. Bei Amyloidose Patienten konnten genetische Varianten auf Chromosom 2 und 8 identifiziert werden, die es wahrscheinliche machen, dass sich das toxische Amyloid im Herzen bzw. in der Leber ablagert.
Kooperationen
DFG Forschergruppe: Mechanismus der Leichtketten-Fehlfaltung bei Patienten mit systemischer Amyloidose
Die Leichtketten-Amyloidose ist eine der häufigsten systemischen Amyloidosen in Deutschland. Bei fortgeschrittenem Nieren- oder Herzbefall kann sie zur raschen Dialyse oder Versterben führen. Die Krankheit beruht auf der Fehlfaltung und Aggregation von Immunoglobulin Antikörper-Leichtketten, welche im Rahmen einer monoklonalen B-Zell-Erkrankung gebildet werden. Eine Besonderheit der AL-Amyloidose ist ihr hochvariables klinisches Erscheinungsbild. Diese Heterogenität ist mutmaßlich die Folge der natürlichen Variabilität von Leichtketten und der polymorphen Aggregatstruktur. Die genauen biochemischen Zusammenhänge sind aber unverstanden. Ziel der Forschergruppe ist es, für zwei besonders prominente klinische Formen der AL-Amyloidose (dominanter Herz- oder Nierenbefall) aufzuklären, wie Krankheitsentstehung und klinisches Erscheinungsbild von spezifischen proteinbiochemischen Eigenschaften abhängen. Die Forschergruppe untersucht hier insbesondere Sequenz und Primärstruktur, proteolytische Prozessierung, Faltung und polymorphe Aggregatstruktur.
Subprojekt : 1 Sequenz Diversität der Leichtketten
PIs Stefan O. Schönland, Stefanie Huhn
Universitätsklinikum Heidelberg; Medizinische Klinik V, Amyloidose Zentrum & Sektion Multiples Myelom
Unser Team
Laborleitung
Stellv. Laborleitung
Projektmanagerin
Technische Assistenten/-innen
Medizinische Dokumentationsassistentin
Ausgewählte Publikationen
- Radamaker L, Lin YH , Annamalai K, Huhn S, Hegenbart U, Schönland SO, Fritz G, Schmidt M, Fändrich M. Cryo-EM structure of a light chain-derived amyloid fibril from a patient with systemic AL amyloidosis. Nat Commun. 2019 Mar 20;10(1):1103.
- Chattopadhyay S, Thomsen H, Yadav P, da Silva Filho MI, Weinhold N, Nöthen MM, Hoffman P, Bertsch U, Huhn S, Morgan GJ, Goldschmidt H, Houlston R, Hemminki K, Försti A. Genome-wide interaction and pathway-based identification of key regulators in multiple myeloma. Commun Biol. 2019.
- Campa D, Martino A, Macauda A, Dudziński M, Suska A, Druzd-Sitek A, Raab MS, Moreno V, Huhn S, Butrym A, Sainz J, Szombath G, Rymko M, Marques H, Lesueur F, Vangsted AJ, Vogel U, Kruszewski M, Subocz E, Buda G, Iskierka-Jażdżewska E, Ríos R, Merz M, Schöttker B, Mazur G, Perrial E, Martinez-Lopez J, Butterbach K, García Sanz R, Goldschmidt H, Brenner H, Jamroziak K, Reis RM, Kadar K, Dumontet C, Wątek M, Haastrup EK, Helbig G, Jurczyszyn A, Jerez A, Varkonyi J, Barington T, Grzasko N, Zaucha JM, Andersen V, Zawirska D, Canzian F. Genetic polymorphisms in genes of class switch recombination and multiple myeloma risk and survival: an IMMEnSE study. Leuk Lymphoma. 2019 Jan 11:1-9.
- Huhn S. ELDA qASO-PCR for High Sensitivity Detection of Tumor Cells in Bone Marrow and Peripheral Blood. Methods Mol Biol. 2018.
- Huhn S, Weinhold N, Nickel J, Pritsch M, Hielscher T, Hummel M, et al. Circulating tumor cells as a biomarker for response to therapy in multiple myeloma patients treated within the GMMG-MM5 trial. Bone Marrow Transplant. 2017.
- da Silva Filho MI, Forsti A, Weinhold N, Meziane I, Campo C, Huhn S, et al. Genome-wide association study of immunoglobulin light chain amyloidosis in three patient cohorts: comparison with myeloma. Leukemia. 2017.
- Meziane I, Huhn S, da Silva Filho MI, Weinhold N, Campo C, Nickel J, et al. Genome-wide association study of clinical parameters in immunoglobulin light chain amyloidosis in three patient cohorts. Haematologica. 2017.
- Weinhold N, Meissner T, Johnson DC, Seckinger A, Moreaux J, Forsti A, Chen B, Nickel J, Chubb D, Rawstron AC et al: The 7p15.3 (rs4487645) association for multiple myeloma shows strong allele-specific regulation of the MYC-interacting gene CDCA7L in malignant plasma cells. Haematologica 2014.
- Campa D, Martino A, Varkonyi J, Lesueur F, Jamroziak K, Landi S, Jurczyszyn A, Marques H, Andersen V, Jurado M et al: Risk of multiple myeloma is associated with polymorphisms within telomerase genes and telomere length. Int J Cancer 2015.
- Weinhold N, Forsti A, da Silva Filho MI, Nickel J, Campo C, Hoffmann P, Nothen MM, Hose D, Goldschmidt H, Jauch A et al: Immunoglobulin light-chain amyloidosis shares genetic susceptibility with multiple myeloma. Leukemia. 2014.
- Weinhold N, Johnson DC, Rawstron AC, Forsti A, Doughty C, Vijayakrishnan J, Broderick P, Dahir NB,Begum DB, Hosking FJ et al: Inherited genetic susceptibility to monoclonal gammopathy of unknownsignificance. Blood 2014.
- Morgan GJ, Johnson DC, Weinhold N, Goldschmidt H, Landgren O, Lynch HT, Hemminki K, Houlston RS: Inherited genetic susceptibility to multiple myeloma. Leukemia. 2014.
- Chubb D, Weinhold N, Broderick P, Chen B, Johnson DC, Forsti A, Vijayakrishnan J, Migliorini G, Dobbins SE, Holroyd A et al: Common variation at 3q26.2, 6p21.33, 17p11.2 and 22q13.1 influences multiple myeloma risk. Nat Genet 2013.
- Weinhold N, Johnson DC, Chubb D, Chen B, Forsti A, Hosking FJ, Broderick P, Ma YP, Dobbins SE, Hose D et al: The CCND1 c.870G>A polymorphism is a risk factor for t(11;14)(q13;q32) multiple myeloma. Nat Genet 2013.
- Martino A, Campa D, Buda G, Sainz J, Garcia-Sanz R, Jamroziak K, Reis RM, Weinhold N, Jurado M, Rios R et al: Polymorphisms in xenobiotic transporters ABCB1, ABCG2, ABCC2, ABCC1, ABCC3 and multiple myeloma risk: a case-control study in the context of the International Multiple Myeloma rESEarch (IMMEnSE) consortium. Leukemia. 2012.
- Campa D, Martino A, Sainz J, Buda G, Jamroziak K, Weinhold N, Vieira Reis RM, Garcia-Sanz R, Jurado M, Rios R et al: Comprehensive investigation of genetic variation in the 8q24 region and multiple myeloma risk in the IMMEnSE consortium. British journal of haematology 2012.
- Broderick P, Chubb D, Johnson DC, Weinhold N, Forsti A, Lloyd A, Olver B, Ma YP, Dobbins SE, Walker BA et al: Common variation at 3p22.1 and 7p15.3 influences multiple myeloma risk. Nat Genet 2012.
- Weinhold N, Moreaux J, Raab MS, Hose D, Hielscher T, Benner A, Meissner T, Ehrbrecht E, Brough M, Jauch A et al: NPM1 is overexpressed in hyperdiploid multiple myeloma due to a gain of chromosome 5 but is not delocalized to the cytoplasm. Genes, chromosomes & cancer 2010.
- Biobank / GMMG Probeneingangslabor
- Myelomregister
- Myelom Studienzentrale / GMMG Studien
- Translationale Myelomforschung
- Funktionelle Bildgebung
- Molekularbiologisches Labor / Studien zur Erblichkeit des Multiplen Myeloms
- Innovative Therapien beim Multiplen Myelom: Phase I, PerMyt, HIPO
- Laboratory for Translational Immunology (TRIM)
- GMMG Assessment Center der Minimalen Resterkrankung (MRD)
Leitung

Dr. sc. hum. Stefanie Huhn
Leitung
(AG Biobank / GMMG Probeneingangslabor / Molekularbiologisches Labor)