DIE NCOUNTER CORE FACILITY
Die nCounter Core Facility wurde 2011 etabliert und ist Teil der CellNetworks Core Technology Platform der Universität Heidelberg und der MULTI-SPACE Plattform der Health & Life Science Alliance.
Die Facility bietet modernste Verfahren für mRNA- und miRNA Expressionsprofiling (nCounter SPRINT Profiler), als auch für Digital Spatial Profiling auf Transkript- (GeoMx Digital Spatial Profiler) und Proteinebene (GeoMx Digital Spatial Profiler, Lunaphore COMET) an. Darüber hinaus gehören verschiedene Analyseverfahren zur Qualitätskontrolle von RNA- und DNA-Proben zum Leistungsspektrum der Facility. Im Forschungsinfrastrukturportal "RIsources" der DFG ist die nCounter Core Facility unter RI_00563 (nCounter SPRINT Profiler, GeoMX Digital Spatial Profiler) und RI_00613 (Lunaphore COMET) gelistet.
DER NCOUNTER SPRINT PROFILER
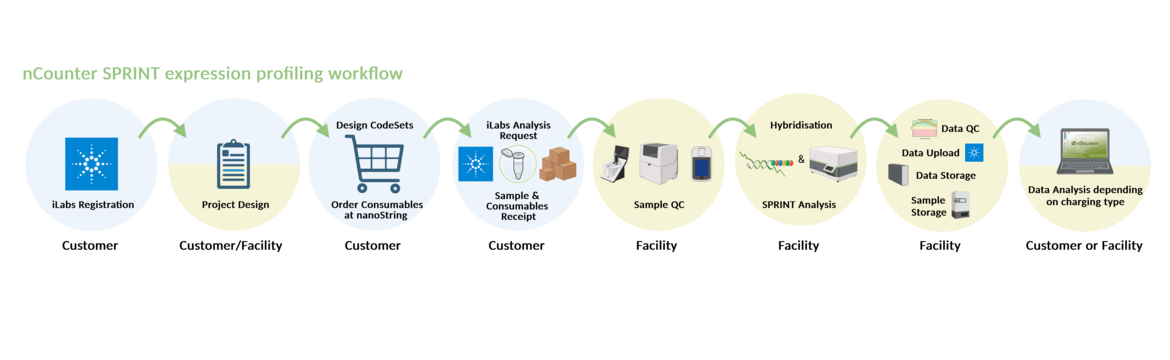
Der nCounter SPRINT Profiler von Nanostring Technologies ist für die digitale Expressionsanalyse von bis zu 800 mRNAs oder miRNAs sowie die CNV-Analyse auf DNA-Ebene geeignet.
Für die Multiplex-Analyse verwendet das Gerät fluoreszenzmarkierte Reportersonden, die aufgrund ihrer Länge von ca. 100 Basen sehr resistent gegen mangelhafte RNA-Qualität sind und sich daher ideal für schwer zu analysierende Proben wie FFPE (Formalin-fixed, paraffin-embedded) Proben eignen.
Die Anwendung einer einzigartigen Kodierungstechnologie ermöglicht die direkte Zählung einzelner RNA-Moleküle auf allen Ebenen der biologischen Expression, mit einer Sensitivität und Spezifität, die mit der Real Time PCR (RT PCR) vergleichbar ist. Darüber hinaus werden für die Analyse nur kleinste Mengen an RNA (25-150ng) oder DNA (150ng) benötigt. Eine Reverse Transkription der RNA ist nicht erforderlich. Der SPRINT Profiler ist ein perfektes System für die Validierung von Microarrays, die Analyse von Signalwegen, die Expressionsanalyse definierter Genlisten (individuell oder vordefiniert) sowie für die Validierung von Biomarkern.
DER GEOMX DIGITAL SPATIAL PROFILER
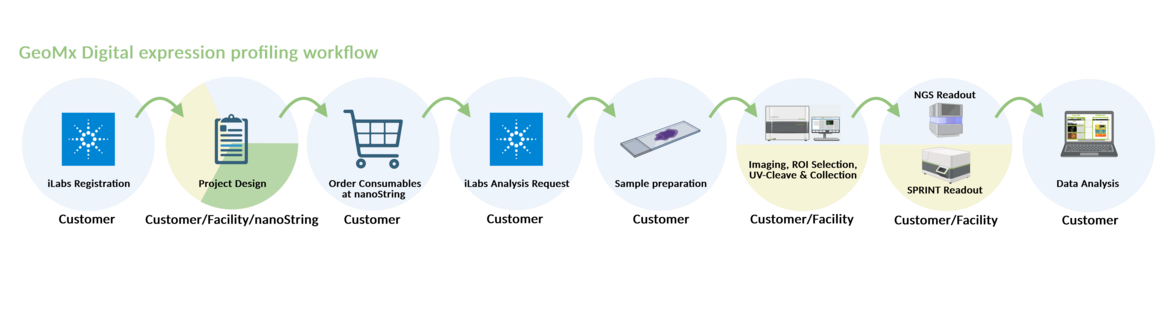
Für die Erstellung von multizellulären Expressionsprofilen auf Transkript- und Proteinebene bietet die nCounter Core Facility den GeoMx Digital Spatial Profiler von Nanostring Technologies an, bei dem an Antikörper oder RNA-Sonden gebundene, durch Licht spaltbare Oligonukleotid-Barcodes verwendet werden, die eine nahezu unbegrenzte Multiplexfähigkeit ermöglichen (~20 000 RNAs, ≥100 Proteine). Außerdem erlaubt das Gerät eine hochkomplexe räumliche Auflösung und Quantifizierung von Proteinen und RNA (Whole Transcriptome Read-out).
LUNAPHORE COMET PLATTFORM
Die Lunaphore COMET Plattform (https://lunaphore.com/products/comet) ermöglicht die simultane Darstellung von bis zu 40 Antikörpern an ein bis vier Gewebeproben gleichzeitig. Der automatisierte Arbeitsablauf und ein präzises Microfluidics-System gewährleisten eine hohe Reproduzierbarkeit. Neben Proteinen können auch spezifische RNA-Proben angefärbt werden.
Die Gewebeproben (FFPE oder Fresh Frozen) werden direkt oder indirekt gefärbt und abgebildet. Der Hintergrund wird automatisiert subtrahiert. Die erzeugten Bilder werden im tiff-Format gespeichert und können damit mit gängigen Programmen ausgewertet werden.
Workflow Lunaphore COMET Plattform:
Nach erfolgter Registrierung bei iLabs und der Anforderung einer „Lunaphore COMET Consultation request“ in iLabs werden das Projekt und die Experimente besprochen. Nach dem Ausfüllen der Anfrage „Lunaphore COMET sample run“ in iLabs und der Zusendung aller benötigten Materialien und Proben werden zunächst Testläufe und anschließend nach weiterer Besprechung die Experimente selbst durchgeführt. Danach können die Daten je nach Absprache abgeholt oder heruntergeladen werden.
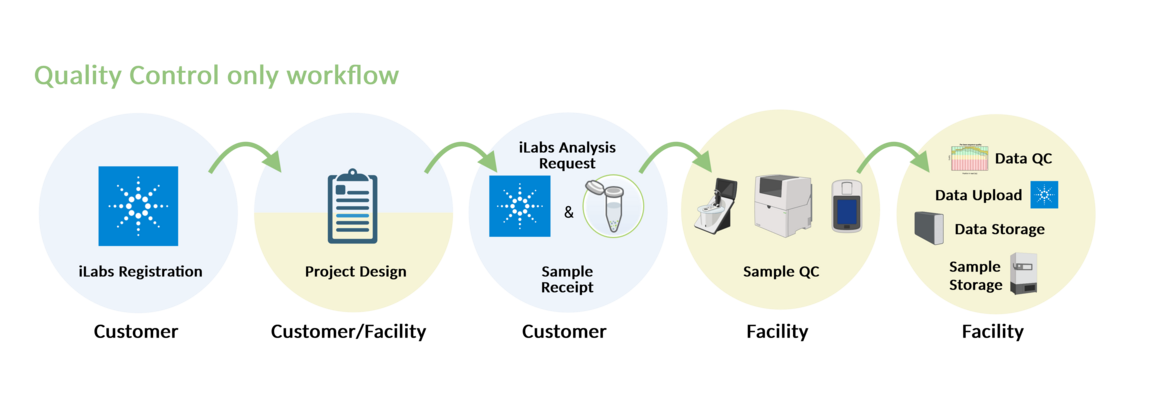
QUALITÄTSKONTROLLE
Für reine Qualitätskontrollanalysen verwenden wir die Agilent TapeStation und das Qubit-System von Thermo Fisher Scientific.