REFERENZLABOR FÜR KLINISCHE STUDIEN
Bedeutung chromosomaler Aberrationen für die Prognose und Pathogenese des Multiplen Myeloms:
Untersuchungen im Rahmen einer multizentrischen Phase III Studie
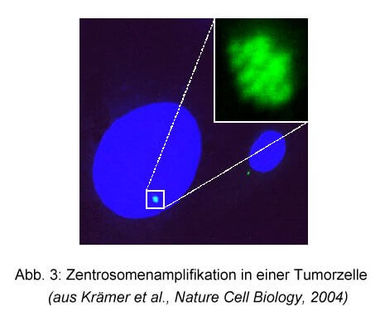
Hintergrund: Das Multiple Myelom (MM) ist eine maligne lymphoproliferative B-Zell-Erkrankung, die durch Vermehrung von monoklonalen Plasmazellen gekennzeichnet ist. Zusätzlich zu den bereits etablierten Prognosefaktoren werden Chromosomenaberrationen als prognostische Parameter diskutiert. In ca. 40% aller MM lassen sich typische Translokationen nachweisen, wodurch die Onkogene (Cyclin D1, FGFR3, MAF, etc) unter die Kontrolle der Enhancer-Elemente des Immunglobulinschwerketten-(IGH)-Locus gelangen. In den übrigen Fällen findet man überwiegend numerische Chromosomenveränderungen. Kürzlich wurden Zentrosomen-Aberrationen als eine mögliche Ursache für Aneuploidieinduktion in hämatologischen Neoplasien beschrieben, die auch beim MM eine Rolle spielen könnten.
Ziele: In dem vom Tumorzentrum Heidelberg/Mannheim geförderten Projekt soll die prognostischen Aussagekraft chromosomaler Veränderungen beim MM prospektiv evaluiert und Zentrosomen-Aberrationen als möglicher Mechanismus zur Entstehung von numerischen Chromosomenaberrationen untersucht werden.
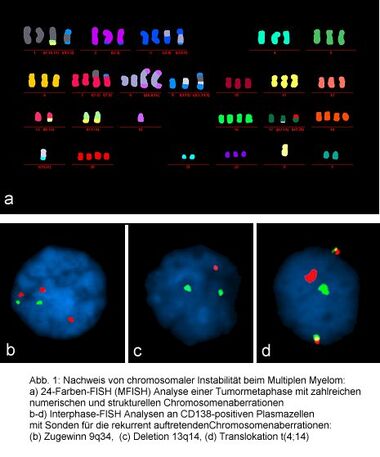
Vorgehen: Mit Hilfe eines MM-spezifischen Sondensatzes zum Nachweis rekurrent auftretender Chromosomenaberrationen werden Interphase-FISH-Analysen an CD138-positiven Plasmazellen aus dem Knochenmark neu diagnostizierter MM-Patienten durchgeführt.
Ergebnisse: Aufgrund von Interphase-FISH-Analysen konnten erstmals bestimmte Chromosomenaberrationen unterschiedlichen MM-Subgruppen zugeordnet und Aberrationen abgegrenzt werden, die erst im späteren Verlauf der Erkrankung und unabhängig von der Subgruppenzugehörigkeit auftreten. Es konnte eine hyperdiploide Subgruppe mit rekurrenten Trisomien und einem günstigen Risikoprofil von einer non-hyperdiploiden Subgruppe mit Translokation t(4;14) häufig in Assoziation mit Deletion 13q14 und einem aggressiven Krankheitsverlauf abgegrenzt werden. Die Subgruppe mit Translokation t(11;14) zeigt eine hohe CCND1-Expression und ein besseres klinisches Outcome während die Subgruppe mit Zugewinn 1q21 mit prognostisch ungünstigen klinischen Parametern assoziiert ist.
Bei Patienten mit Multiplem Myelom konnte gezeigt werden, dass der Nachweis einer Deletion 17p13 und/oder einer Translokation t(4;14)(p16;q32.3) bzw. t(14;16)(q32.3;q23) mit einer ungünstigen Prognose assoziiert ist (ca. 25% aller neudiagnostizierten MM-Patienten). Die Deletionen 8p21 und 13q14 werden häufig assoziiert mit den o. g. Hochrisiko-Aberrationen beobachtet. Verschiedene Studien zeigen jedoch, dass die Deletion 13q14 alleine nicht mit einer ungünstigen Prognose verbunden sind. Aktuelle Berichte deuten darauf hin, dass Zugewinne von Chromosom 1q21 mit einer Krankheitsprogression assoziiert sind, wobei insbesondere Patienten mit einem Zugewinn 1q21 von >3 Kopien eine besonders schlechte Prognose zeigen. Der Nachweis einer Translokation t(11;14)(q13;q32.3) weist bei Patienten, die mit einer Hochdosis-Therapie und autologer Blutstammzell-Transplantation behandelt werden, auf einen eher günstigen Verlauf hin. Zugewinne von 5p15/5q35, 9q34, 15q22, 11q22.3 und 19q13 zählen zu den häufigen Veränderungen beim Multiplen Myelom und sind mit einem hyperdiploiden Karyotyp assoziiert und sprechen ebenfalls für einen eher günstigen Krankheitsverlauf.
Bei Patienten mit asymptomatischen SMM konnte gezeigt werden, dass eine Deletion 17p13, die Translokation t(4;14) und ein Zugewinn 1q21 sowie ein hyperdiploider Karyotyp (charakterisiert durch mindestens zwei Zugewinne der Chromosomen 5, 9, 15 und 19) mit einem erhöhten Progressionsrisiko assoziiert ist (Neben et al. 2013, J Clin Oncol, Rajkumar et al, Leukemia 2013).
Kooperationspartner:
PD Dr. med. Dipl-Phys Dirk Hose, Leiter des Labors für Myelomforschung, Medizinische Klinik V, Universitätsklinikum Heidelberg.
Prof. Dr. med. Jens Hillengaß, Leiter der autologen Transplantations-Ambulanz, Stellvertretender Leiter der Sektion Multiples Myelom, Leitung der Arbeitsgruppe "Hämatoonkologische Bildgebung", Medizinische Klinik V, Universitätsklinikum Heidelberg.
PD Dr. med. Marc-Steffen Raab, Leitung der Max-Eder Gruppe "Experimentelle Therapien hämatologischer Neoplasien" und der klinischen Arbeitsgruppe "Innovative Myelomtherapie", Medizinische Klinik V, Universitätsklinikum Heidelberg.
Ausgewählte Publikationen
- Neben K, Ott G, Schweizer S, Kalla J, Tews B, Katzenberger T, Hahn M, Rosenwald A, Ho AD, Müller-Hermelink HK, Lichter L, Krämer A. (2007). Expression of centrosome-associated gene products is linked to tetraploidization in mantle cell lymphoma. Int J Cancer, 120:1669-1677.
- Hensel M, Zoz M, Giesecke C, Benner A, Neben K, Jauch A, Stilgenbauer S, Ho AD, Krämer A. (2007). Centrosome aberrations correlate with cytogenetic and clinical risk profile and time to first treatment in B-cell chronic lymphocytic leukaemia. Int J Cancer, 121:978.983.
- Cremer FW, Kartal M, Hose D, Bila J, Buck I, Bellos F, Raab MS, Brough M, Moebus A, Hager HD, Goldschmidt H, Moos M, Bartram CR, Jauch A. (2005a). High incidence and intraclonal heterogeneity of chromosome 11 aberrations in patients with newly diagnosed multiple myeloma detected by multiprobe interphase FISH. Cancer Genet Cytogenet, 161:116-24.
- Cremer FW, Bila J, Buck I, Kartal M, Hose D, Ittrich C, Benner A, Raab MS, Theil AC, Moos M, Goldschmidt H, Bartram CR, Jauch A. (2005b). Delineation of distinct subgroups of multiple myeloma and a model for clonal evolution based on interphase cytogenetics. Genes Chromosomes Cancer, 44:194-203.
- Moreaux J, Cremer FW, Reme T, Raab M, Mahtouk K, Kaukel P, Pantesco V, DE Vos J, Jourdan E, Jauch A, Legouffe E, Moos M, Fiol G, Goldschmidt H, Rossi JF, Hose D, Klein B (2005). The level of TACI gene expression in myeloma cells is associated with a signature of microenvironment dependence versus a plasmablastic signature. Blood 106: 1021-1030
- Krämer A., Neben K, Ho AD. (2005). Centrosome aberrations in hematological malignancies. Cell Biol Int, 29:375-383.
- Neben K, Tews B, Wrobel G, Hahn M, Kokocinski F, Giesecke C, Krause U, Ho AD, Krämer A, Lichter P. (2004). Gene expression patterns in acute myeloid leukemia correlate with centrosome aberrations and numerical chromosome changes. Oncogene, 23: 2379-84.
- Neben K, Giesecke C, Schweizer S, Ho AD, Krämer A. (2003). Centrosome aberrations in acute myeloid leukemia are correlated with cytogenetic risk profile. Blood, 101:289-91.
- Krämer A, Schweizer S, Neben K, Giesecke C, Kalla J, Katzenberger T, Benner A, Muller-Hermelink HK, Ho AD, Ott G. (2003). Centrosome aberrations as a possible mechanism for chromosomal instability in non-Hodgkin’s Lymphoma. Leukemia, 17:2207-2213.